Rで RandomForest を試してみたのでメモ。
参考書籍は, はじめてのパターン認識です。
決定木
決定木は木構造の有向グラフです。目的変数が量的データの場合は回帰木となり, 質的データの場合は分類木となります。
Rでは {rpart} で決定木を構築できます。
アンサンブル法
アンサンブル法は, 複数の弱分類器から高性能な強学習器を生成する仕組みで集団学習とも言います。
アンサンブル法はブースティングとバギングの2つの枠組みがよく知られています。
バギング
バギング (Bootstrap AGGregatING) はブートストラップサンプリング (Bootstrap Sampling) と集約 (Aggregating) の2つの要素からなります。
ブートストラップサンプリングは n 個のサンプルデータから重複を許してランダムに n 個選択するリサンプリング手法です。
得られたサブセットで弱学習器を構築します。これを繰り返し複数の弱学習器の出力を多数決や平均化などで集約し最終的な出力とします。
Random Forest
Random Forest は決定木を弱学習器に用いたバギングに加えてランダムな特徴選択を取り入れている点が大きな特徴です。
弱学習器 (決定木) はエントロピーやジニ不純度を基準としノードを作ります。指定した深さに到達するか末端に達するまで再帰的に繰り返します。
Random Forest の特徴として以下が挙げられます。
- 非線形分離可能で精度が高い
- 特徴量が高次元でも効率的に学習できる
- 説明力の高い説明変数の数に比べノイズとなる説明変数の数が多いと精度が下がる
- 弱学習器の生成は並列化が容易
また, bagging によりサブセットを作る時のランダム性によって誤ったラベルが少し混ざっていても, 最終的には弱学習器の集約になるので出力に影響しにくくノイズに強いという特徴もあります。
R で Random Forest
{randomForest} パッケージは論文の著者による R での実装です。randomForest() で指定できる主なパラメータは以下です。
- y: 目的変数。 気をつける点は factor 型の場合に分類, それ以外は回帰が適用される。
- ntree: 決定木の数。小さすぎると選択されない入力が発生する可能性がある。デフォルトで 500
- mtry: ランダムに選択される特徴数。デフォルトでは分類では k の対数値, 回帰では k/3
- nodesize: 末端ノード (terminal node) の最小サイズ。分類では 1, 回帰では 5
- importance: 変数重要度を評価するか
- proximity: 入力間の近傍性を計算するか
今回は比較として {rpart} を用います。
require(rpart)
require(randomForest)
require(RColorBrewer)
set.seed(1500)
# random sampling
n <- nrow(iris)
s <- sample(n, n * 0.5)
iris.train <- iris[s,]
iris.test <- iris[-s,]
model <- randomForest(
Species ~ .,
data = iris.train,
ntree = 500,
proximity = TRUE
)
pred.model <- predict(
model,
newdata = iris.test,
type = 'class'
)
table(pred.model, iris.test[,5])
getTree(model, 1, labelVar = TRUE)
importance(model)
# MeanDecreaseGini
# Sepal.Length 3.590923
# Sepal.Width 2.366265
# Petal.Length 21.485794
# Petal.Width 20.524631
# MDG
varImpPlot(model)
MDSplot(model, iris$Species, palette = rep(1, 3), pch = as.numeric(iris$Species))
split.screen(c(2,1))
split.screen(c(1,3), screen = 2)
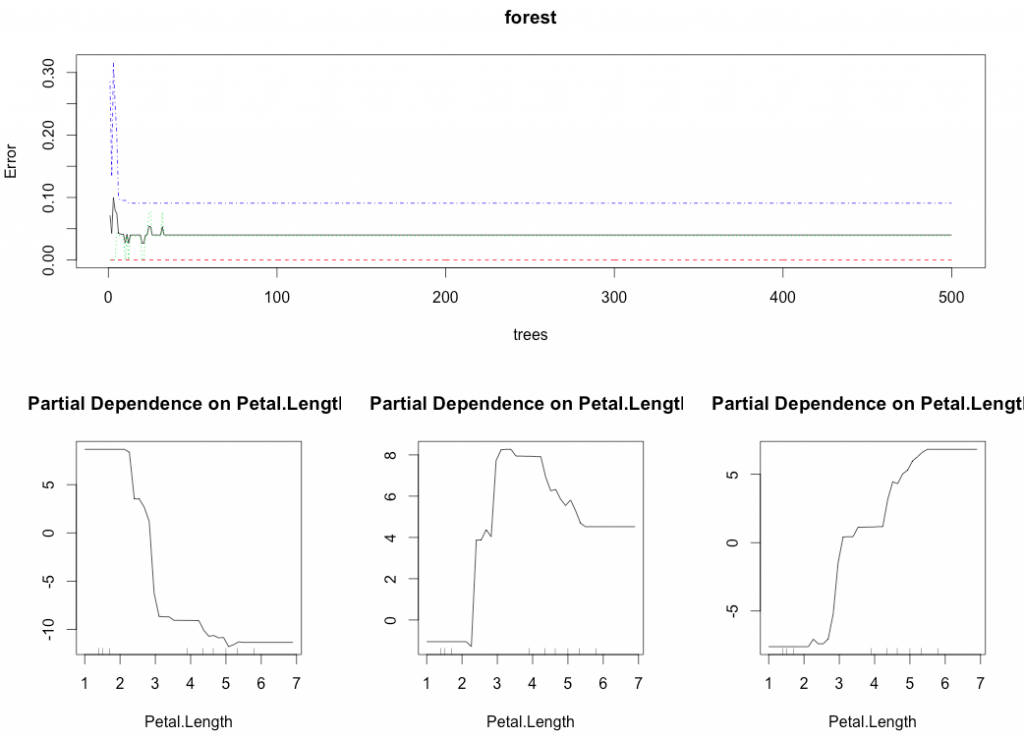
screen(3); partialPlot(model, iris, Petal.Length, 'setosa')
screen(4); partialPlot(model, iris, Petal.Length, 'versicolor')
screen(5); partialPlot(model, iris, Petal.Length, 'virginica')
split.screen(c(2,1), screen = 1)
screen(1); plot(model)
close.screen(all=T)
# decision tree
tree <- rpart(
Species ~ .,
data = iris.train
)
pred.tree <- predict(
tree,
iris.test,
type = 'class'
)
{rpart} より僅かにテストデータでの正解率が高くなりました。
> table(pred.model, iris.test[,5])
pred.model setosa versicolor virginica
setosa 30 0 0
versicolor 0 16 2
virginica 0 1 26
> table(pred.rpart, iris.test[,5])
pred.tree setosa versicolor virginica
setosa 30 0 0
versicolor 0 14 1
virginica 0 3 27
getTree() で生成されたモデルの内容, varImpPlot() で生成したモデルにおける変数重要度を表示することができます。
Rの code は GitHub にあります。
{randomForest} 以外の R の Random Forest 実装は {ranger}, {Rborist} などがあるそうです。[2]
[1] R言語によるランダムフォレスト徹底入門
[2] 最近のRのランダムフォレストパッケージ -ranger/Rborist-